Design av FRET-TaqMan-sonder för multiplex realtids-PCR med användning av en intern positiv kontroll
introduktion
realtids-PCR möjliggör kontinuerlig övervakning av fluorofor-fluorescens under genereringen av PCR-produkter i ett slutet rörformat. För närvarande använder tillgängliga metoder antingen märkta prober eller DNA-interkalerande färgämne för att övervaka förstärkningen av PCR-produkten. Taqman-sonden är en enkelsträngad oligonukleotid innehållande en fluorofor och släckare placerad 10-30 baser från varandra. Taqman-sonden hydrolyseras under förstärkningen på grund av den 5′-3′ dubbelsträngsspecifika exonukleasaktiviteten hos Taq-polymeraset som separerar fluoroforen från släckaren, vilket resulterar i fluorescensökning. Realtids PCR-instrument är utrustade med fluorescensdetektorer och programvara som kan uppskatta cykeltröskeln (Ct), cykeln vid vilken fluorescens är större än bakgrundsfluorescens, för positiva reaktioner. Tekniken kan emellertid inte skilja ett sant negativt resultat från ett falskt negativt när PCR påverkas av förstärkningshämmare. Efter nukleinsyraextraktion rapporteras att hämmare fortfarande kan förekomma från kliniska prover (t.ex. hemoglobin), miljöprover (t. ex. humussyror och fulvinsyror) och kemikalier som används vid nukleinsyraextraktion (t. ex. etanol, tvättmedel eller chaotropa medel). Tillförlitligheten hos diagnostiska analyser ökar genom införandet av en intern kontroll nukleinsyra som kan indikera närvaron och effekten av PCR-hämmare (1,2). En intern positiv kontroll (IPC) förstärks samtidigt i närvaro av en målsekvens med användning av en märkt fluorofor som avger ljus vid en annan våglängd än den fluorofor som används för målsekvensanalysen, med de två fluoroforerna detekterade i olika kanaler av realtids-PCR-instrumentet. Den vanliga interna kontrollen för PCR är en plasmid som innehåller en sekvens som liknar analysmålet förutom sondregionen. Ett begränsat antal IPC-molekyler läggs till individuellt analysmål och samförstärks med målnukleinsyran; således är en positiv IPC-signal bevis på att förstärkningsreaktionen fortsatte tillräckligt för att generera en positiv signal från mycket små mängder målnukleinsyra. Denna funktion är viktig för att säkerställa ekvivalent förstärkning av IPC och målnukleinsyran (3-9). Vissa enheter – som LightCycler 1.2 och LightCycler 2 (Roche Applied Science, Indianapolis, i, USA), den robusta avancerade Patogenidentifieringsanordningen (R. A. P. I. D.) instrument (Idaho Technology, Salt Lake City, UT, USA) och handhållna PCR-instrument i realtid—är endast utrustade med en ljuskälla och tillhörande utsläppskanal. Multiplexering på dessa typer av instrument kräver användning av multimärkta oligonukleotider och fluorescensresonansenergiöverföring (FRET)-konceptet. Emellertid kan FRET-sonder vara utmanande att designa och kan sakna robusthet i hypervariabla regioner och när icke-optimala förstärkningsförhållanden är närvarande. Användningen av FRET-sonder kräver identifiering av två regioner för två sonder. FRET-sondsystemet är beroende av intilliggande hybridisering av oligonukleotider hos de två enkelmärkta proberna på ett sådant sätt detekteras fluorescensen hos acceptorfärgämnet genom excitering av fluorofor fäst vid den andra sonden. En stor nackdel med FRET-sonden är kravet på ett större sekvensområde som är nödvändigt för att rymma två intilliggande sonder med ett gap på 2-5 nukleotider. Möjligheten finns för multiplexering med andra etikettsystem, såsom MegaStokes dye (DYXL; Dyomics, Jena, Tyskland) och Pulsar 650 (Biosearch Technologies, Inc., Novato, CA, USA), men dessa märkningssystem kräver spektral information för att kompensera för kanalövergång och är inte allmänt tillgängliga. FRET-TaqMan-sonden som utvecklats i denna studie kräver emellertid inte färgkompensationsprogramvara och använder fluoroforer (FAM, Cy5.5) som är allmänt tillgängliga. Denna rapport fokuserar på utformningen av en trippelmärkt sond för att möjliggöra multiplexering i instrument utrustade med endast en blå lysdiod (LED) excitationskälla (470 nm) och tillhörande tre motsvarande detekteringskanaler (t.ex. 530 nm, 640 nm och 705 nm).
material och metoder
Sonddesignprincip
alla experiment utfördes på Idaho Technologies R. A. P. I. D.-system, ett 32-prov portabelt PCR-instrument i realtid utrustat med en blå LED för excitation (470 nm) och associerade tre fluorescensdetekteringskanaler som mäter fluorescens vid 530, 640 och 705 nm för mono-och dubbelfärgsanalyser. Primers JVAF och JVAR (10) och sond 5′-FAM/BHQ-1 syntetiserades vid CDC Biotechnology Core Facility (Atlanta, GA, USA) och sond 5 ’fam-Cy5.5/3’ BHQ-3 syntetiserades vid Idaho Technology. För att möjliggöra duplex Taqman PCR märktes en sond med 5 ’-FAM/BHQ-1 (520 nm fluorescensvåglängd) och en annan med 5 ’fam-Cy5.5/3’ BHQ-3 (705 nm fluorescensvåglängd). I lösning förblir sonden märkt med 5 ’fam-Cy5.5/3’ BHQ-3 släckt vid hybridisering till en sekvens, och vid efterföljande klyvning av sonden genom enzymatisk primerförlängning elimineras släckaren. Detta resulterar i FRET genom excitering av FAM av ljuskällan av R. A. P. I. D. instrumentet. Excitationsenergin överförs till acceptorfluoroforen, Cy5.5, och den emitterade fluorescensen mäts i en andra fluorescenskanal (fluorescenskanal F3 för R. A. P. I. D. instrument). Utsläppen av CY5.5 fluorescens är resultatet av kombinationen av FRET-och TaqMan-sondtekniker, så dessa trippelmärkta sonder kan kallas ”FRET-TaqMan” – sonder.
Primers, prober och plasmid
för att demonstrera multiplexeringsförmågan hos FRET-TaqMan-designen använde vi en genusspecifik Cryptosporidium realtids PCR-analys för att förstärka en 159-bp-del av 18S rRNA-genen (10). Den främre primern (JVAF) var 5′-ATGACGGGTAACGGGGAAT-3′, omvänd primer (JVAR) var 5′-CCAATTACAAAAC-CAAAAAGTCC-3’och TaqMan-sonden (JVAP) var 5′-FAM-CGCGCCTGCT-GCCTTCCTTAGATG–BHQ1-3′, motsvarande nukleotider 100-118, 258-236 och 184-161, av genbank anslutning nr. AY458612. Primers för konstruktionen av Cryptosporidium IPC innehöll samma uppsättning primersekvenser rapporterade för förstärkning av Cryptosporidium spp., med undantag för sondbindningsområdet för mål-DNA, som ersattes med den artificiella sekvensen 5′-TTGTGCTCGTAGTCGCCTCTCTCTCTCTCTCTCTCT-3′ (JPIPC). Denna sondsekvens hade en glödgningstemperatur på 69 CCC och var utformad för att inte ha någon sekundär struktur. Teoretisk specificitet för intern sond fastställdes genom en sprängning (11) sökning av sonden och primersekvenserna där ingen exakt matchning hittades med någon kryptosporidium-sekvens. IPC-fragmentet syntetiserades och klonades till en plasmid genom integrerad DNA-teknik (Coralville, IA, USA). En känd koncentration av plasmid-DNA (100 kopior/reaktion) tillsattes i masterblandningen av alla reaktioner utom i negativ kontroll. En trippelmärkt sond (JPIPC) som är specifik för IPC syntetiserades med Idaho-teknik, som visas i Figur 1. Fam/Cy5.5 / BHQ3 oligo-nukleotidproben syntetiserades från och med 3′-änden med icke-fluorescerande Svart Hålsläckare (BHQ-3) kontrollerat porglas (CPG). 5 ’- änden konjugerades sedan med en CY5.5-fosporamidit följt av en terminal 5’ fluorescein (6-FAM) fosporamidit.
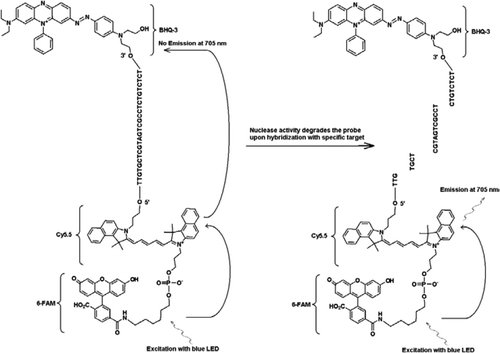
FRET-TaqMan-sonden innehåller tre etiketter: en svart hålsläckare i slutet av 3′ – armen, en emitterfluorofor (Cy5.5) och en skördare fluorofor (FAM) sammanfogade i slutet av sin 5′ – arm. FAM absorberar effektivt energi från Blå LED som ljuskälla. I avsaknad av mål är sonden mörk eftersom den energi som absorberas av FAM överförs till Cy5.5 och i sin tur den energi som frigörs från Cy5.5 överförs till släckaren och går förlorad som värmeenergi. I närvaro av mål klyvs Taqman-sonden av aktiviteten av Taq-polymeras och quencher frigörs och den energi som absorberas av FAM överförs till Cy5.5 genom FRET-mekanismen för att avge fluorescens vid 705 nm.
realtids PCR-analys
flödescytometri sorterade Cryptosporidium parvum oocyster (200 oocyster per rör) erhållna från University of Wisconsin State Laboratory of Hygiene (Madison, WI, USA). DNA från C. parvum oocysts extraherades med användning av ett protokoll som beskrivs av Hill et al. (12) och upphängd i 80 ci l Tris EDTA (te, pH 8.0) buffert. Två mikro liter DNA tillsattes per reaktion, vilket motsvarade 5 oocyster (motsvarande 100 kopior av mål-DNA). Förstärkningsreaktionsblandningen bestod av Kvantifast sond PCR + ROX kit reaktionsblandning (Cat. no. 204354; Qiagen) utan ROX tillagd, JVAF, JVAR primers (0,4 crimm vardera), JVAP-sond (0,2 crimm), JPIPC-sond (0,2 crimm) och 100 kopior av IPC. En alikvot (2 mikrosekl) av det extraherade DNA-provet tillsattes till reaktionskapillären (Roche Diagnostics, Indianapolis, IN, USA) innehållande 18 mikrosekl-reaktionsblandning. En C. parvum genomisk DNA-positiv kontroll (Figur 2, plot 1), plasmid IPC (Figur 2, plot 2), en blandning av C. parvum positiv kontroll och IPC (Figur 2, plot 3) och en no-Mall kontroll (NTC) bestående av nukleasfritt vatten (Cat. Nej. AM9932; Ambion, Foster City, CA, USA) istället för DNA (Figur 2, plot 4) inkluderades i varje experiment. Protokollet tog 30 min att slutföra med följande PCR-tillstånd: hot-start denatureringssteg vid 95 c c i 3 min, följt av 45 cykler med en 95 c c denaturering för 5 s, 60 C C glödgning för 30 s med enstaka fluorescensförvärv (med en temperaturramphastighet på 20 c/s). Ett positivt resultat registrerades för FAM i Kanal F1 (530 nm) och för Cy5.5 i Kanal F3 (705 nm). Förstärkningsreaktioner utfördes i tre exemplar. Amplikoner (159 bp) visualiserades genom agarosgelelektrofores och Glödgrön nukleinsyrafärgning (eenzym, Gaithersburg, MD, USA) för att bekräfta PCR-produktens specificitet.
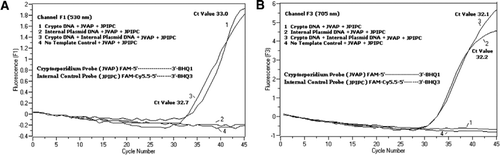
en ekvimolär blandning av en FAM / BHQ1 och en FAM/Cy5.5/BHQ3 tillsattes i alla PCR-reaktionsblandningar. (A) kanal F1 monitorer ökning av fam fluorescens för närvaron av C. parvum. Plot 1 (kanal F1 samlar främst FAM-signal) innehållande endast C. parvum DNA avslöjade kryptosporidium-specifik FAM-signal i PCR. Plot 2 innehöll endast plasmid-DNA och ingen FAM-signal från FAM/Cy5.5/BHQ3-sond. Plot 3 innehöll både C. parvum och plasmid DNA men producerade endast C. parvum-specifik FAM-signal. Plot 4 var ett negativt prov innehållande endast nukleasfritt vatten i stället för något mall-DNA; ingen fluorescens observerades från sonderna. (B) kanal F3 monitorer ökning av Cy5.5 för förekomst av intern kontroll plasmid DNA. Plot 1 innehöll endast C. parvum DNA och ingen fluorescenssignal från FAM/ BHQ1-sond. Plot 2 (kanal F3 samlar främst Cy5. 5-signal) innehållande endast plasmid–DNA avslöjade intern plasmidkontroll (IPC) – specifik Cy5.5-signal i PCR. Plot 3 innehöll både C. parvum och plasmid DNA men producerade endast IPC-specifik Cy5. 5-signal. Plot 4 var ett negativt prov innehållande endast nukleasfritt vatten i stället för något mall-DNA; ingen fluorescens observerades från sonderna.
resultat och diskussion
realtids PCR fluorescenssignaler som återspeglar förstärkning av C. parvum genomiskt DNA detekterades i F1-kanalen (figur 2a) på grund av fam-märkning av TaqMan-sonden. Fluorescenssignal som återspeglar förstärkning av IPC detekterades i F3-kanalen (Figur 2B) på grund av FAM/Cy5.5 märkning av IPC TaqMan-sonden. Denna sond producerade en ökning av Cy5. 5 i F3-kanalen (Cy5.5), men ingen ökning av FAM i F1-kanalen (FAM) när BHQ3 separeras från FAM/Cy5.5 på grund av nukleasaktivitet, eftersom energi överfördes från FAM till Cy5.5. På liknande sätt detekterades i duplexreaktionen C. parvum genomisk DNA-amplifiering i F1-kanalen, medan IPC-förstärkningen samtidigt detekterades i F3-kanalen. FAM har en excitation vid 495 nm och en emission vid 520 nm. Cy5. 5 har en 690 nm excitation och en 705 nm emission. Med sådan närhet mellan Cy5.5 och FAM på den trippelmärkta IPC-sonden avger CY5.5-fluoroforn fluorescens på grund av fullständig överföring av energi från FAM-fluoroforn, vilket resulterar i ingen fluorescensutsläpp av FAM i F1-kanalen (figur 2a). Negativa kontrollreaktioner visade ingen ökning av fluorescens på grund av FAM eller Cy5.5. Dessa resultat visar också att en enda blå (470 nm) LED-källa är tillräcklig för att övervaka dubbla fluorescensutsläpp genom F1 (530 nm) och F3 (705 nm) kanaler utan spektral överlappning i respektive övervakningskanaler för FAM och Cy5.5. FRET-TaqMan-tekniken testades också med användning av en panel med 12 DNA-extrakt från avföringsprover (8 från prover som tidigare bestämts vara positiva för Cryptosporidium spp. och fyra exemplar som var negativa för Cryptosporidium spp.). 18S rRNA-analysen kunde upptäcka alla åtta Cryptosporidium spp. IPC Taqman-analysen visade sig förstärka endast IPC-plasmid-DNA. IPC-analysen resulterade i Ct-värden på 32-37, vilket indikerade upp till 5 Ct-värden för PCR-hämning för vissa avföringsprover (32 var det förväntade Ct-värdet för inga hämningsreaktioner).
den nuvarande tekniken skiljer sig från traditionellt använt FRET genom att en fluorescerande märkt oligonukleotidsond används (FRET-Taqman-sond) istället för två sonder (FRET-sonder) där en sond innehåller donatorfluoroforen och den andra sonden innehåller acceptorfluoroforen (13,14). FRET-TaqMan-sonden är märkt vid 5 ’- änden med två fluoroforer av donator (FAM) och acceptor (Cy5.5) fluoroforer och vid 3′-änden med en BHQ-3-släckare som en TaqMan-sond. Den unika sondbindande regionen väljs för att skilja sig från mål-DNA för att övervaka två olika fluorescensmätningar i två olika detekteringskanaler (530 nm i F1 och 705 nm i F3) från samma förstärkningsreaktion utan att behöva inkludera färgkompensationsprogramvara. Mål-DNA och IPC hade samma primerbindningsställen men en annan sondsekvens. Detta säkerställer att användningen av samma primers för båda DNA-målen för att minimera primer-dimerbildning eller interaktion mellan primeruppsättningar. När fam / Cy5.5 fluoroforer placeras nära vid 5’ – änden sker resonansenergiöverföring med en ökning av fluorescensutsläpp av CY5.5 fluorofor. Användningen av BHQ3, en icke-fluorescensacceptor för Cy5.5, minskar emellertid potentiell bakgrundsfluorescens, vilket resulterar i ett högre signal-brusförhållande. Det visade sig att tillägg av 100 kopior av IPC till en reaktion innehållande 100 kopior av genomisk C. parvum DNA resulterade i liknande Ct-värden (Ct = 32) när Cryptosporidium TaqMan-analysen utfördes som en singleplex-analys och som en duplexanalys med IPC TaqMan-analysen. När det applicerades på en panel av kryptosporidium-positiva DNA-prover indikerade IPC att vissa prover inte innehöll PCR-hämmare (baserat på förväntade Ct-värden på 32), men andra prover var associerade med hämning av upp till 5 Ct-värden.
den nyutvecklade interna PCR-positiva kontrollen visade sig vara känslig och specifik för samtidig detektering av två mål i samma reaktion på grund av märkningen med FAM/Cy5.5-BHQ3 och FAM/BHQ1 för respektive IPC-och 18S rRNA-målgen i en dubbelfärgad multiplex PCR för övervakning i F1-och F3-fluorescenskanaler i R. A. P. I. D-instrumentet. Användningen av 5 ’fam-Cy5.5/3’ BHQ-3–märkt sond som den andra reportern är fördelaktig eftersom emissionsmaxima för de två sonderna var väl åtskilda från varandra. Den TRIPPELMÄRKTA sonddesignen FRET-TaqMan kan ha applikationer för att utveckla en billig, disponibel” lab-on-a-chip ” – enhet med optik bestående av en blå LED-excitation och två utsläppskällor på 530 nm och 705 nm, med lämpliga optiska filter för att detektera FAM i en kanal och Cy5.5 i den andra. Denna kombination skulle inte kräva två högdrivna fluorescenskällor och detekteringssystem.
erkännanden
författarna erkänner hjälp av Mike Powers vid DNA-sekvensering och genomisk kärnanläggning (University of Utah, Salt Lake City, UT, USA) och Rachelle Muller vid Idaho Technology, Inc. för diskussion om utvecklingen av den interna positiva kontrollen för R. A. P. I. D-cyklaren. Denna publikation stöddes delvis av medel som gjordes tillgängliga via Centers for Disease Control and Prevention, Coordinating Office for Terrorism Preparedness and Emergency Response. Användning av handelsnamn och kommersiella källor är endast för identifiering och innebär inte godkännande av Centers for Disease Control and Prevention eller US Department of Health and Human Services.
- 1. Hartman, L. J., S. R. Coyne och D. A. Norwood. 2005. Utveckling av en ny intern positiv kontroll för Taqman-baserade analyser. Mol. Cell. Sonder 19: 51-59.Crossref, Medline, CAS, Google Scholar
- 2. Hoorfar, J., B. Malorny, A. Abdulmawjood, N. Cook, M. Wagner och P. Fach. 2004. Praktiska överväganden vid utformning av interna förstärkningskontroller för diagnostiska PCR-analyser. J. Clin. Mikrobiol. 42:1863–1868.Crossref, Medline, CAS, Google Scholar
- 3. Betsou, F., K. Beaumont, J. M. Sueur och J. Orfila. 2003. Konstruktion och utvärdering av intern kontroll DNA för PCR-förstärkning av Chlamydia trachomatis DNA från urinprover. J. Clin. Mikrobiol. 41:1274–1276.Crossref, Medline, CAS, Google Scholar
- 4. Burggraf, S. och B. Olgemoller. 2004. Enkel teknik för intern kontroll av realtidsförstärkningsanalyser. Clin. Chem. 50:819–825.Crossref, Medline, CAS, Google Scholar
- 5. Drosten, C., M. Weber, E. Seifried och W. K. Roth. 2000. Utvärdering av en ny PCR-analys med konkurrenskraftig intern kontrollsekvens för screening av blodgivare. Transfusion 40: 718-724.Crossref, Medline, CAS, Google Scholar
- 6. Gruber, F., F. G. Falkner, F. Dorner och T. Hammerle. 2001. Kvantifiering av viralt DNA genom realtids-PCR som applicerar duplexförstärkning, intern standardisering och tvåfärgsfluorescensdetektering. Appl. Miljö. Mikrobiol. 67:2837–2839.Crossref, Medline, CAS, Google Scholar
- 7. Rosenstraus, M., Z. Wang, S. Y. Chang, D. DeBonville och J. P. Spadoro. 1998. En intern kontroll för rutindiagnostisk PCR: design, Egenskaper och effekt på klinisk prestanda. J. Clin. Mikrobiol. 36:191–197.Medline, CAS, Google Scholar
- 8. Stocher, M., V. Leb och J. Berg. 2003. Ett bekvämt tillvägagångssätt för generering av multipel intern kontroll DNA för en panel av realtids PCR-analyser. J. Virol. Metoder 108: 1-8.Crossref, Medline, CAS, Google Scholar
- 9. Zimmermann, K. och J. W. Mannhalter. 1996. Tekniska aspekter av kvantitativ konkurrenskraftig PCR. Bioteknik 21: 268-279.Länk, CAS, Google Scholar
- 10. Jothikumar, N., A. J. da Silva, I. Moura, Y. Qvarnstrom och V. R. Hill. 2008. Detektion och differentiering av Cryptosporidium hominis och Cryptosporidium parvum genom dubbla Taqman-analyser. J. Med. Mikrobiol. 57:1099–1105.Crossref, Medline, CAS, Google Scholar
- 11. Altschul, S. F., W. Gish, W. Miller, E. W. Myers och D. J. Lipman. 1990. Grundläggande lokal anpassning sökverktyg. J. Mol. Biol. 215:403–410.Crossref, Medline, CAS, Google Scholar
- 12. Hill, V. R., A. M. Kahler, N. Jothikumar, T. B. Johnson, D. Hahn och T. L. Cromeans. 2007. Multistate utvärdering av ett ultrafiltreringsbaserat förfarande för samtidig återvinning av enteriska mikrober i 100-liters kranvattenprover. Appl. Miljö. Mikrobiol. 73:4218–4225.Crossref, Medline, CAS, Google Scholar
- 13. Bernard, P. S., M. J. Lay och C. T. Wittwer. 1998. Integrerad förstärkning och detektion av C677T-punktmutationen i metylentetrahydrofolatreduktasgenen genom fluorescensresonansenergiöverföring och sondsmältningskurvor. Anal. Biochem. 255:101–107.Crossref, Medline, CAS, Google Scholar
- 14. Wittwer, C. T., M. G. Herrmann, A. A. Moss och R. P. Rasmussen. 1997. Continuous fluorescence monitoring of rapid cycle DNA amplification. BioTechniques 22:130–138.Link, CAS, Google Scholar