A concepção de sondas FRET-TaqMan para PCR multiplex em tempo real utilizando um controlo interno positivo
introdução
PCR em tempo Real permite a monitorização contínua da fluorescência fluoróforica durante a geração de produtos PCR em formato de tubo fechado. Atualmente, os métodos disponíveis utilizam sondas rotuladas ou corante INTERCALANTE de DNA para monitorar a amplificação do produto PCR. A sonda TaqMan é um oligonucleótido de cadeia simples contendo um fluoróforo e quencher colocados entre 10-30 bases. A sonda TaqMan é hidrolisada durante a amplificação devido à atividade de exonuclease específica de 5′-3′ da Taq polimerase que separa o fluoróforo do aquecedor, resultando em aumento de fluorescência. Os instrumentos de PCR em tempo Real estão equipados com detectores de fluorescência e software capaz de estimar o limiar do ciclo (Ct), o ciclo em que a fluorescência é maior do que a fluorescência de fundo, para reações positivas. No entanto, a técnica não pode diferenciar um resultado negativo verdadeiro de um falso negativo quando a PCR é afetada por inibidores de amplificação. Após a extração de ácido nucleico, é relatado que os inibidores ainda podem estar presentes a partir de amostras clínicas (por exemplo, hemoglobina), amostras ambientais (por exemplo, ácidos úmico e fulvico), e produtos químicos empregados durante a extração de ácido nucleico (por exemplo, etanol, detergentes ou agentes chaotrópicos). A fiabilidade dos ensaios de diagnóstico é aumentada pela inclusão de um ácido nucleico de controlo interno que possa indicar a presença e o impacto dos inibidores da PCR (1,2). Um controlo interno positivo (IPC) é amplificado simultaneamente na presença de uma sequência-alvo utilizando um fluoróforo rotulado que emite luz a um comprimento de onda diferente do fluoróforo utilizado para o doseamento da sequência-alvo, com os dois fluoróforos detectados em canais diferentes pelo instrumento PCR em tempo real. O controlo interno normalmente utilizado para a PCR é um plasmídeo que contém uma sequência semelhante à do alvo do ensaio, excepto para a região da sonda. Um número limitado de moléculas IPC são adicionadas ao alvo do ensaio individual e co-amplificadas com o ácido nucleico alvo; assim, um sinal positivo de IPC é a evidência de que a reação de amplificação procedeu suficientemente para gerar um sinal positivo a partir de quantidades muito pequenas de ácido nucleico alvo. Esta característica é importante para garantir a amplificação equivalente do IPC e do ácido nucleico alvo (3-9). No entanto, alguns dispositivos—como o LightCycler 1.2 e LightCycler 2 (Roche Applied Science, Indianapolis, IN, USA), o Ruggeded Advanced Pathogen Identification Device (R. A. P. I. D.) instrument (Idaho Technology, Salt Lake City, UT, USA), and handheld real-time PCR instruments—are only equipped with one light source and associated emission channel. A multiplexação desses tipos de instrumentos requer o uso de oligonucleótidos multi-rotulados e o conceito de transferência de energia de ressonância de fluorescência (FRET). No entanto, as sondas FRET podem ser um desafio para o design e podem não ter robustez em regiões hipervariáveis e quando as condições de amplificação não ótimas estão presentes. O uso de sondas de traste requer a identificação de duas regiões para duas sondas. O sistema de sonda FRET baseia-se na hibridação adjacente de oligonucleótidos das duas sondas com um único rótulo de tal forma que a fluorescência do corante aceitador é detectada através da excitação do fluoróforo ligado à segunda sonda. Uma grande desvantagem da sonda FRET é a exigência de uma maior área de sequência necessária para acomodar duas sondas adjacentes com um intervalo de 2-5 nucleótidos. Existe a possibilidade de multiplexagem com outros sistemas de etiquetas, como o corante MegaStokes (DYXL; Dyomics, Jena, Alemanha) e o Pulsar 650 (Biosearch Technologies, Inc., Novato, CA, EUA), mas estes sistemas de rotulagem requerem informação espectral para compensar o canal crosstalk e não estão amplamente disponíveis. A sonda FRET-TaqMan desenvolvida neste estudo, no entanto, não requer software de compensação de cores e usa fluoróforos (FAM, Cy5.5) que estão amplamente disponíveis. O presente relatório centra-se no projeto de uma sonda tripla etiquetada para permitir a multiplexação em instrumentos equipados com apenas uma fonte de excitação de díodo emissor de luz azul (LED) (470 nm) e associados três canais de detecção correspondentes (por exemplo, 530 nm, 640 nm e 705 nm).todos os experimentos foram realizados no sistema R. A. P. I. D. da Idaho Technology, um aparelho portátil PCR portátil de 32 amostras em tempo real equipado com um LED azul para excitação (470 nm) e três canais de detecção de fluorescência associados que medem fluorescência a 530, 640 e 705 nm para testes mono e duais. Primers JVAF and JVAR (10) and probe 5′-FAM/BHQ-1 were synthesized at the CDC Biotechnology Core Facility (Atlanta, GA, USA) and probe 5’FAM-Cy5.5/3’BHQ-3 was synthesized at Idaho Technology. Para permitir o duplex TaqMan PCR, uma sonda foi rotulada com 5′-FAM/BHQ-1 (comprimento de onda de fluorescência de 520 nm) e outra com 5’FAM-Cy5.5/3’bhq-3 (comprimento de onda de fluorescência de 705 nm). Em solução, a sonda rotulada com 5’FAM-Cy5.5/3’bhq-3 permanece apagada após hibridação para uma sequência, e após a clivagem subsequente da sonda por extensão enzimática do primer, o aquecedor é eliminado. Isso resulta em FRET através da excitação do FAM pela fonte de luz do instrumento R. A. P. I. D. A energia de excitação é transferida para o receptor fluoróforo, Cy5.5, e a fluorescência emitida é medida num segundo canal de fluorescência (canal de fluorescência F3 para o instrumento R. A. P. I. D.). A emissão de fluorescência de Cy5. 5 é o resultado da combinação de técnicas de sonda de FRET e TaqMan, de modo que estas sondas triplamente rotuladas podem ser referidas como sondas de “FRET-TaqMan”.
Primers, sondas e plasmídeos
para demonstrar as capacidades de multiplexagem do projeto FRET-TaqMan, usámos um teste de Cryptosporidium em tempo real específico do género para amplificar uma porção de 159-bp do gene rRNA 18S (10). O forward primer (JVAF) foi de 5′-ATGACGGGTAACGGGGAAT-3′, reverse primer (JVAR) foi de 5′-CCAATTACAAAAC-CAAAAAGTCC-3′, e sonda TaqMan (JVAP) foi de 5′-FAM-CGCGCCTGCT-GCCTTCCTTAGATG-BHQ1–3′, correspondente ao nucleotídeos 100-118, 258-236, e 184-161, respectivamente, do GenBank adesão não. AY458612. Os iniciadores para a construção do Cryptosporidium IPC continham o mesmo conjunto de sequências de iniciadores relatadas para amplificação de Cryptosporidium spp., excepto para a região de ligação à sonda do ADN-alvo, que foi substituído pela sequência artificial 5′-TTGCTCGTAGTCGCCTCTCTCTCTCTCTCTCTCTCTCT-3′ (JPIPC). Esta sequência de sonda tinha uma temperatura de recozimento de 69 ° C e foi projetada para não ter estrutura secundária. A especificidade teórica para a sonda interna foi determinada por uma busca BLAST (11) da sonda e sequências primer nas quais não foi encontrada nenhuma correspondência exata com qualquer sequência criptosporidium. O fragmento IPC foi sintetizado e clonado em um plasmídeo por tecnologias integradas de DNA (Coralville, IA, EUA). Foi adicionada uma concentração conhecida de ADN plasmídeo (100 cópias/reacção) na mistura principal de todas as reacções, excepto no controlo negativo. A triple-labeled probe (JPIPC) specific to the IPC was synthesized by Idaho Technology, as shown in Figure 1. A sonda FAM/Cy5.5/BHQ3 oligo-nucleotídeo foi sintetizada a partir da extremidade 3′ com aquecedor não-fluorescente de Buraco Negro (BHQ-3) controlado por por poros glass (CPG). A extremidade 5′ foi então conjugada com uma fosforamidite Cy5.5 seguida de uma fluoresceína 5 terminal (6-FAM).
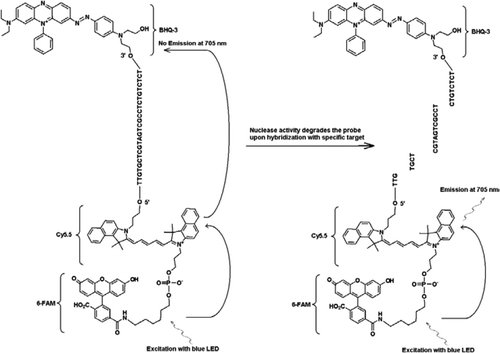
A sonda FRET-TaqMan contém três Etiquetas: uma fracção de quebra-buracos negros no final do braço de 3′, um emissor fluoróforo (Cy5.5), e um colector de fluoróforo (FAM) unidos no final do braço de 5′. A FAM absorve eficientemente a energia do LED azul como uma fonte de luz. Na ausência de alvos, a sonda é escura porque a energia absorvida pelo FAM é transferida para o Cy5.5 e, por sua vez, a energia liberada do Cy5.5 é transferido para o aquecedor e é perdido como energia de calor. Na presença de alvos, a sonda TaqMan é clivada pela atividade da Taq polimerase e aqucher é liberada e a energia absorvida pelo FAM é transferida para Cy5.5 através do mecanismo FRET para emitir fluorescência a 705 nm.
Real-ensaio de PCR em tempo
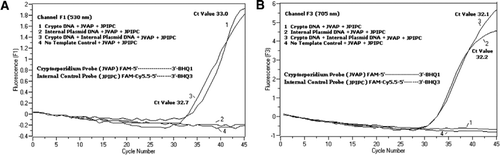
citometria de Fluxo classificados Cryptosporidium parvum oocistos (200 oocistos por tubo) obtido a partir da Universidade de Wisconsin, Estado do Laboratório de Higiene (Madison, WI, EUA). ADN de C. os oocistos de parvum foram extraídos usando um protocolo descrito por Hill et al. (12) e suspenso em 80 µL de tampão Tris EDTA (TE, pH 8.0). Foram adicionados dois micro-litros de ADN por reacção, correspondendo a 5 oocistos (equivalente a 100 cópias do ADN-alvo). A mistura de reacção de amplificação consistiu na mistura de reacção de Quantifast sonda PCR + ROX kit (Cat. n. o 204354; Qiagen) sem adição de ROX, jvaf, iniciadores JVAR (0,4 µM cada), sonda JVAP (0,2 µM), sonda JPIPC (0,2 µM) e 100 cópias da IPC. Uma alíquota (2 µL) da amostra de DNA extraída foi adicionada ao capilar de reação (Roche Diagnostics, Indianapolis, IN, USA) contendo 18 µL de mistura de reação. Um controlo positivo do ADN genómico de C. parvum( Figura 2, Parcela 1), o plasmídeo IPC (Figura 2, Parcela 2), uma mistura do controlo positivo de C. parvum e do IPC (Figura 2, Parcela 3), e um controlo sem modelo (NTC) constituído por água sem nucleases (Cat. Não. AM9932; Ambion, Foster City, CA, EUA) em vez do DNA (Figura 2, parcela 4) foram incluídos em cada experiência. O protocolo demorou ∼30 min para completar com as seguintes condições PCR: etapa de desnaturação com arranque a quente a 95°C durante 3 minutos, seguida de 45 ciclos com uma desnaturação de 95°C durante 5 s, 60°C recozendo durante 30 S com uma única aquisição de fluorescência (com uma taxa de rampa de temperatura de 20°C/s). Um resultado positivo foi gravado para FAM no canal F1 (530 nm) e para Cy5.5 no canal F3 (705 nm). As reacções de amplificação foram realizadas em triplicado. Os amplificadores (159 bp) foram visualizados pela electroforese em gel de agarose e pela coloração do ácido nucleico Verde-clarão (Eenzyme, Gaithersburg, MD, EUA) para confirmar a especificidade do produto PCR.
resultados e discussão
sinais de fluorescência da PCR em tempo Real reflectindo a amplificação do ADN genómico de C. parvum foi detectado no canal F1 (figura 2A) devido à rotulagem da FAM da sonda TaqMan. Foi detectado um sinal de fluorescência reflectindo a amplificação da IPC no canal F3 (figura 2B) devido ao FAM/Cy5.5 etiquetagem da sonda IPC TaqMan. Esta sonda produziu-se um aumento na Cy5.5 no canal F3 (Cy5.5), mas sem aumento no FAM na F1 canal (FAM) quando BHQ3 é separado do FAM/Cy5.5 devido a atividade de nuclease, porque a energia foi transferida da FAM Cy5.5. Similarmente, na reação duplex, a amplificação do DNA genômico de C. parvum foi detectada no canal F1, enquanto a amplificação IPC foi detectada simultaneamente no canal F3. O FAM tem uma excitação a 495 nm e uma emissão a 520 nm. O Cy5. 5 tem uma excitação de 690 nm e uma emissão de 705 nm. Com uma proximidade tão estreita entre o Cy5.5 e o FAM na sonda tripla IPC, o fluoróforo Cy5.5 emite fluorescência devido à transferência completa de energia do fluoróforo FAM, não resultando na emissão de fluorescência do FAM no canal F1 (figura 2A). As reacções de controlo negativas não revelaram um aumento da fluorescência devido à FAM ou ao Cy5.5. Estes resultados também demonstram que uma única fonte LED Azul (470 nm) é suficiente para monitorar as emissões duais de fluorescência através de canais F1 (530 nm) e F3 (705 nm) sem qualquer sobreposição espectral nos respectivos canais de monitorização para FAM e Cy5.5. A técnica FRET-TaqMan também foi testada usando um painel de 12 extratos de DNA de amostras de fezes (8 de amostras previamente determinadas como positivas para Cryptosporidium spp. e quatro espécimes que deram negativo para Cryptosporidium spp.). O ensaio rRNA 18S foi capaz de detectar todos os oito Cryptosporidium spp. O teste IPC TaqMan foi encontrado para amplificar apenas o DNA plasmídeo IPC. O ensaio IPC resultou em valores Ct de 32-37, que indicaram até 5 valores Ct de inibição da PCR para algumas amostras de fezes (32 Foi o valor CT esperado para reacções sem inibição).
A presente técnica é diferente da tradicionalmente utilizada TRASTE em que um microesferas rotulado oligonucleotide sonda é utilizada (TRASTE-sonda TaqMan) em vez de duas sondas (TRASTE sondas) em que uma sonda contém o fluoróforo doador e a outra sonda contém o acceptor fluoróforo (13,14). A sonda FRET-TaqMan é rotulada na extremidade 5′ com dois fluoróforos de doadores (FAM) e aceitadores (Cy5.5) fluoróforos e na extremidade 3′ com um quencher BHQ-3 como uma sonda TaqMan. Seleciona-se a região única de ligação à sonda para diferenciar do ADN-alvo, de modo a monitorizar duas medições de fluorescência diferentes em dois canais de detecção diferentes (530 nm em F1 e 705 nm em F3) da mesma reação de amplificação sem a necessidade de incluir software de compensação de cores. O ADN-alvo e o IPC tinham os mesmos locais de ligação, mas uma sequência de sonda diferente. Isto garante que o uso dos mesmos iniciadores para ambos os alvos de DNA para minimizar a formação de primer-dimer ou interação entre os conjuntos de primer. Quando os fluoróforos FAM / Cy5.5 são colocados de perto na extremidade de 5′, a transferência de energia de ressonância ocorre com um aumento na emissão de fluorescência do fluoróforo Cy5.5. No entanto, o uso de BHQ3, um aceitador de não fluorescência para Cy5.5, diminui a potencial fluorescência de fundo, o que resulta em uma maior razão sinal-ruído. Descobriu-se que a adição de 100 cópias do IPC a uma reação contendo ∼100 cópias do genomic C. o ADN parvum resultou em valores de Ct semelhantes (Ct = 32) quando o ensaio Cryptosporidium TaqMan foi realizado como um ensaio singleplex e como um ensaio duplex com o ensaio IPC TaqMan. Quando aplicado a um painel de amostras de ADN positivo de Cryptosporidium, a IPC indicou que algumas amostras não continham inibidores PCR (com base nos valores de Ct esperados de 32), mas outras amostras estavam associadas à inibição de até 5 valores de Ct.
A recém-desenvolvida interno PCR de controlo positivo provou ser sensível e específico para detecção simultânea de dois alvos na mesma reação, devido à etiquetagem com o FAM/Cy5.5-BHQ3 e FAM/BHQ1 para o respectivo IPC e 18S rRNA gene alvo em uma dupla-cor PCR multiplex para o monitoramento em F1 e F3 canais de fluorescência da R. A. P. I. D instrumento. O uso da sonda 5’FAM-Cy5.5/3’bhq-3–rotulado como o segundo repórter é vantajoso porque os máximos de emissão das duas sondas foram bem espaçados um do outro. The FRET-TaqMan triple-labeled probe design could have applications in developing an inexpensive, disposable “lab-on-a-chip” device with optics consisting of one blue LED excitation and two emission sources of 530 nm and 705 nm, with appropriate optical filters to detect FAM in one channel and Cy5.5 in the other. Esta combinação não exigiria dois sistemas de fonte de fluorescência de alta potência e de detecção.
agradecimentos
os autores reconhecem a assistência de Mike Powers na sequenciação de DNA e no núcleo genômico (Universidade de Utah, Salt Lake City, UT, EUA) e Rachelle Muller na Idaho Technology, Inc. for discussion on the development of the internal positive control for the R. A. P. I. D cycler. Esta publicação foi apoiada, em parte, por Fundos disponibilizados através dos Centros De Controle e prevenção de doenças, escritório de Coordenação de Preparação para o terrorismo e Resposta de emergência. O uso de nomes comerciais e fontes comerciais é apenas para identificação e não implica endosso pelos Centros de controle e prevenção de doenças ou pelo Departamento de Saúde e Serviços Humanos dos EUA.
- 1. Hartman, L. J., S. R. Coyne, and D. A. Norwood. 2005. Desenvolvimento de um novo controle interno positivo para testes baseados em Taqman. Mol. Celula. Sondas 19: 51-59.Crossref, Medline, CAS, Google Scholar
- 2. Hoorfar, J., B. Malorny, A. Abdulmawwood, N. Cook, M. Wagner, and P. Fach. 2004. Considerações práticas na concepção dos controlos de amplificação interna para os ensaios de diagnóstico por PCR. J. Clin. Microbiol. 42:1863–1868.Crossref, Medline, CAS, Google Scholar
- 3. Betsou, F., K. Beaumont, J. M. Sueur, and J. Orfila. 2003. Construction and evaluation of internal control DNA for PCR amplification of Chlamydia trachomatis DNA from urine samples. J. Clin. Microbiol. 41:1274–1276.Crossref, Medline, CAS, Google Scholar
- 4. Burggraf, S. E B. Olgemoller. 2004. Técnica simples para o controlo interno dos ensaios de amplificação em tempo real. Clin. Chem. 50:819–825.Crossref, Medline, CAS, Google Scholar
- 5. Drosten, C., M. Weber, E. Seifried, and W. K. Roth. 2000. Avaliação de um novo ensaio PCR com sequência de controlo interno competitiva para rastreio de dadores de sangue. Transfusão 40: 718-724.Crossref, Medline, CAS, Google Scholar
- 6. Gruber, F., F. G. Falkner, F. Dorner, and T. Hammerle. 2001. Quantificação do DNA viral por PCR em tempo real aplicando amplificação duplex, padronização interna e detecção de fluorescência de duas cores. Appl. Ambiente. Microbiol. 67:2837–2839.Crossref, Medline, CAS, Google Scholar
- 7. Rosenstraus, M., Z. Wang, S. Y. Chang, D. DeBonville, and J. P. Spadoro. 1998. Um controlo interno para a PCR de diagnóstico de rotina: concepção, propriedades e efeito no desempenho clínico. J. Clin. Microbiol. 36:191–197.Medline, CAS, Google Scholar
- 8. Stocher, M., V. Leb, and J. Berg. 2003. Uma abordagem conveniente para a geração de múltiplos DNA de controle interno para um painel de testes de PCR em tempo real. J. Virol. Methods 108: 1-8.Crossref, Medline, CAS, Google Scholar
- 9. Zimmermann, K. and J. W. Mannhalter. 1996. Aspectos técnicos da PCR competitiva quantitativa. BioTechniques 21: 268-279.Link, CAS, Google Scholar
- 10. Jothikumar, N., A. J. Da Silva, I. Moura, Y. Qvarnstrom, and V. R. Hill. 2008. Detecção e diferenciação de Cryptosporidium hominis e Cryptosporidium parvum por testes duais de TaqMan. J. Med. Microbiol. 57:1099–1105.Crossref, Medline, CAS, Google Scholar
- 11. Altschul, S. F., W. Gish, W. Miller, E. W. Myers, e D. J. Lipman. 1990. Ferramenta básica de pesquisa de alinhamento local. J. Mol. Biol. 215:403–410.Crossref, Medline, CAS, Google Scholar
- 12. Hill, V. R., A. M. Kahler, N. Jothikumar, T. B. Johnson, D. Hahn, e T. L. Cromeans. 2007. Avaliação por vários estados de um procedimento de ultrafiltração para a recuperação simultânea de micróbios entéricos em amostras de água da torneira de 100 litros. Appl. Ambiente. Microbiol. 73:4218–4225.Crossref, Medline, CAS, Google Scholar
- 13. Bernard, P. S., M. J. Lay, and C. T. Wittwer. 1998. Amplificação integrada e detecção da mutação do ponto C677T no gene da metilenetetra-hidrofolato redutase por transferência de energia por ressonância de fluorescência e curvas de fusão de sonda. Anal. Bioquímica. 255:101–107.Crossref, Medline, CAS, Google Scholar
- 14. Wittwer, C. T., M. G. Herrmann, A. A. Moss, e R. P. Rasmussen. 1997. Continuous fluorescence monitoring of rapid cycle DNA amplification. BioTechniques 22:130–138.Link, CAS, Google Scholar