Progettazione di sonde FRET-TaqMan per PCR multiplex real-time con controllo positivo interno
Introduzione
La PCR real-time consente il monitoraggio continuo della fluorescenza dei fluorofori durante la generazione di prodotti PCR in formato a tubo chiuso. Attualmente, i metodi disponibili utilizzano sonde etichettate o colorante intercalante del DNA per monitorare l’amplificazione del prodotto PCR. La sonda TaqMan è un oligonucleotide a filamento singolo contenente un fluoroforo e un quencher posti a 10-30 basi a parte. La sonda TaqMan viene idrolizzata durante l’amplificazione a causa dell’attività esonucleasica specifica a doppio filamento 5′-3′ della polimerasi Taq che separa il fluoroforo dal quencher, con conseguente aumento della fluorescenza. Gli strumenti di PCR in tempo reale sono dotati di rivelatori di fluorescenza e software in grado di stimare la soglia di ciclo (Ct), il ciclo in cui la fluorescenza è maggiore della fluorescenza di fondo, per reazioni positive. Tuttavia, la tecnica non può differenziare un risultato negativo vero da un falso negativo quando la PCR è influenzata dagli inibitori di amplificazione. Dopo l’estrazione dell’acido nucleico, è stato riferito che gli inibitori possono essere ancora presenti da campioni clinici (ad esempio, emoglobina), campioni ambientali (ad esempio, acidi umici e fulvici) e sostanze chimiche impiegate durante l’estrazione dell’acido nucleico (ad esempio, etanolo, detergenti o agenti chaotropici). L’affidabilità dei test diagnostici è aumentata dall’inclusione di un acido nucleico di controllo interno che può indicare la presenza e l’impatto degli inibitori della PCR (1,2). Un controllo positivo interno (IPC) viene amplificato simultaneamente in presenza di una sequenza target utilizzando un fluoroforo marcato che emette luce a una lunghezza d’onda diversa rispetto al fluoroforo utilizzato per il test della sequenza target, con i due fluorofori rilevati in canali diversi dallo strumento PCR in tempo reale. Il controllo interno comunemente usato per PCR è un plasmide che contiene una sequenza simile a quella dell’obiettivo di analisi ad eccezione della regione della sonda. Un numero limitato di molecole IPC viene aggiunto al singolo bersaglio di analisi e co-amplificato con l’acido nucleico bersaglio; pertanto, un segnale IPC positivo è la prova che la reazione di amplificazione ha proceduto sufficientemente per generare un segnale positivo da quantità molto piccole di acido nucleico bersaglio. Questa caratteristica è importante per garantire un’amplificazione equivalente dell’IPC e dell’acido nucleico bersaglio (3-9). Tuttavia, alcuni dispositivi-come il LightCycler 1.2 e LightCycler 2 (Roche Applied Science, Indianapolis, IN, USA), il Ruggedized Advanced Pathogen Identification Device (R. A. P. I. D.) strumento (Idaho Technology, Salt Lake City, UT, USA), e palmare in tempo reale PCR strumenti-sono dotati solo di una sorgente luminosa e canale di emissione associato. Multiplexing su questi tipi di strumenti richiede l’uso di oligonucleotidi multi-etichettati e il concetto di trasferimento di energia di risonanza di fluorescenza (FRET). Tuttavia, le sonde FRET possono essere difficili da progettare e possono mancare di robustezza nelle regioni ipervariabili e quando sono presenti condizioni di amplificazione non ottimali. L’uso di sonde FRET richiede l’identificazione di due regioni per due sonde. Il sistema di sonda FRET si basa sull’ibridazione adiacente di oligonucleotidi delle due sonde a marchio singolo in modo tale che la fluorescenza del colorante accettore venga rilevata attraverso l’eccitazione del fluoroforo collegato alla seconda sonda. Uno svantaggio principale della sonda FRET è il requisito di un’area di sequenza più ampia necessaria per ospitare due sonde adiacenti con uno spazio di 2-5 nucleotidi. Esiste la possibilità di multiplexare con altri sistemi di etichette, come MegaStokes dye (DYXL; Dyomics, Jena, Germania) e Pulsar 650 (Biosearch Technologies, Inc., Novato, CA, USA), ma questi sistemi di etichettatura richiedono informazioni spettrali per compensare la diafonia del canale e non sono ampiamente disponibili. La sonda FRET-TaqMan sviluppata in questo studio, tuttavia, non richiede software di compensazione del colore e utilizza fluorofori (FAM, Cy5.5) che sono ampiamente disponibili. Il presente rapporto si concentra sulla progettazione di una sonda a tripla etichetta per consentire il multiplexing in strumenti dotati di una sola sorgente di eccitazione a diodi emettitori di luce blu (LED) (470 nm) e associati tre canali di rilevamento corrispondenti (ad esempio, 530 nm, 640 nm e 705 nm).
Materiali e metodi
Sonda principio di progettazione
Tutti gli esperimenti sono stati eseguiti su Idaho Tecnologia R. A. P. I. D. sistema, 32-esempio di portatile in tempo reale di PCR strumento dotato di un LED blu per l’eccitazione (470 nm) e associati tre rilevazione a fluorescenza canali che misura la fluorescenza a 530, 640, e 705 nm per mono-e dual-color dosaggi. I primer JVAF e JVAR (10) e probe 5′-FAM/BHQ-1 sono stati sintetizzati presso la CDC Biotechnology Core Facility (Atlanta, GA, USA) e probe 5’FAM-Cy5.5/3’BHQ-3 è stato sintetizzato presso Idaho Technology. Per abilitare la PCR TAQMAN duplex, una sonda è stata etichettata con 5′-FAM/BHQ-1 (lunghezza d’onda di fluorescenza di 520 nm) e un’altra con 5’FAM-Cy5.5/3’BHQ-3 (lunghezza d’onda di fluorescenza di 705 nm). In soluzione, la sonda etichettata con 5’FAM-Cy5.5/3’BHQ-3 rimane estiguuta su ibridazione ad una sequenza e sulla scissione successiva della sonda dall’estensione enzimatica dell’iniettore, il quencher è eliminato. Ciò si traduce in FRET attraverso l’eccitazione di FAM dalla sorgente luminosa dello strumento R. A. P. I. D. L’energia di eccitazione viene trasferita al fluoroforo accettore, Cy5.5, e la fluorescenza emessa viene misurata in un secondo canale di fluorescenza (canale di fluorescenza F3 per lo strumento R. A. P. I. D.). L’emissione di fluorescenza Cy5.5 è il risultato della combinazione di tecniche di sonda FRET e TaqMan, quindi queste sonde a tripla etichetta possono essere denominate sonde “FRET-TaqMan”.
Primer, sonde e plasmidi
Per dimostrare le capacità di multiplazione del progetto FRET-TaqMan, abbiamo utilizzato un test PCR in tempo reale di Cryptosporidium specifico per genere per amplificare una porzione di 159 bp del gene 18S rRNA (10). Il primer forward (JVAF) era 5′-ATGACGGGTAACGGGGAAT-3′, il primer reverse (JVAR) era 5′-CCAATTACAAAAC-CAAAAAGTCC-3′ e la sonda TaqMan (JVAP) era 5′-FAM-CGCGCCTGCT-GCCTTCCTTAGATG-BHQ1–3′, corrispondente ai nucleotidi 100-118, 258-236, e 184-161, rispettivamente, di adesione GenBank n. AY458612. I primer per la costruzione del Cryptosporidium IPC contenevano lo stesso set di sequenze di primer riportate per l’amplificazione di Cryptosporidium spp., fatta eccezione per la regione di legame della sonda del DNA bersaglio, che è stata sostituita con la sequenza artificiale 5 ‘- TTGTGCTCGTAGTCGCCTCTGTCTCT-3 ‘ (JPIPC). Questa sequenza di sonda aveva una temperatura di ricottura di 69°C ed è stata progettata per non avere alcuna struttura secondaria. La specificità teorica per la sonda interna è stata accertata da una ricerca BLAST (11) della sonda e delle sequenze di primer in cui non è stata trovata alcuna corrispondenza esatta con alcuna sequenza di Cryptosporidium. Il frammento IPC è stato sintetizzato e clonato in un plasmide da Integrated DNA Technologies (Coralville, IA, USA). Una concentrazione nota di DNA plasmidico (100 copie/reazione) è stata aggiunta nel mix principale di tutte le reazioni tranne che nel controllo negativo. Una sonda a tripla etichetta (JPIPC) specifica per l’IPC è stata sintetizzata dalla tecnologia Idaho, come mostrato in Figura 1. La sonda oligo-nucleotide FAM/Cy5.5/BHQ3 è stata sintetizzata a partire dall’estremità 3’ con vetro poro controllato (CPG) non fluorescente Black Hole Quencher (BHQ-3). L’estremità 5 ‘è stata quindi coniugata con una phosporamidite Cy5.5 seguita da una phosporamidite 5’ fluoresceina (6-FAM) terminale.
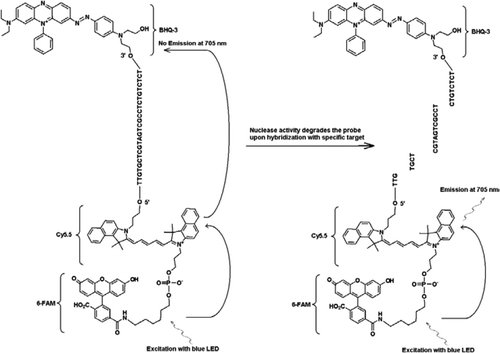
La sonda FRET-TaqMan contiene tre etichette: una porzione di quencher buco nero alla fine del braccio 3′, un fluoroforo emettitore (Cy5.5), e un fluoroforo harvester (FAM) uniti insieme alla fine del suo braccio 5′. Il FAM assorbe in modo efficiente l’energia dal LED blu come fonte di luce. In assenza di bersagli, la sonda è scura perché l’energia assorbita dalla FAM viene trasferita a Cy5.5 e, a sua volta, l’energia rilasciata da Cy5.5 viene trasferito al quencher e viene perso come energia termica. In presenza di bersagli, la sonda TaqMan viene scissa dall’attività della polimerasi Taq e quencher viene rilasciato e l’energia assorbita dalla FAM viene trasferita a Cy5.5 attraverso il meccanismo FRET per emettere fluorescenza a 705 nm.
Analisi PCR in tempo reale
La citometria a flusso ha ordinato oocisti di Cryptosporidium parvum (200 oocisti per tubo) ottenuti dal laboratorio di igiene statale dell’Università del Wisconsin (Madison, WI, USA). DNA da C. oocisti parvum è stato estratto utilizzando un protocollo descritto da Hill et al. (12) e sospeso in tampone Tris EDTA (TE, pH 8,0) da 80 µL. Sono stati aggiunti due micro-litri di DNA per reazione, che corrispondevano a 5 oocisti (equivalenti a 100 copie di DNA bersaglio). La miscela di reazione di amplificazione consisteva in Quantifast Sonda PCR + ROX kit miscela di reazione (Cat. no. 204354; Qiagen) senza ROX aggiunto, JVAF, primer JVAR (0,4 µM ciascuno), sonda JVAP (0,2 µM), sonda JPIPC (0,2 µM) e 100 copie dell’IPC. Un’aliquota (2 µL) del campione di DNA estratto è stata aggiunta al capillare di reazione (Roche Diagnostics, Indianapolis, IN, USA) contenente una miscela di reazione da 18 µL. Un controllo positivo del DNA genomico di C. parvum (Figura 2, diagramma 1), l’IPC del plasmide (Figura 2, diagramma 2), una miscela del controllo positivo di C. parvum e dell’IPC (Figura 2, diagramma 3) e un controllo senza modello (NTC) che consiste dell’acqua nucleasi-libera (Cat. No. AM9932; Ambion, Foster City, CA, USA) invece del DNA (Figura 2, trama 4) sono stati inclusi in ogni esperimento. Il protocollo ha richiesto ∼30 min per essere completato con le seguenti condizioni di PCR: fase di denaturazione a caldo a 95 ° C per 3 min, seguita da 45 cicli con denaturazione a 95 ° C per 5 s, ricottura a 60°C per 30 s con acquisizione a fluorescenza singola (con una velocità di rampa di temperatura di 20°C/s). Un risultato positivo è stato registrato per FAM nel canale F1 (530 nm) e per Cy5.5 nel canale F3 (705 nm). Le reazioni di amplificazione sono state effettuate in triplice copia. Gli ampliconi (159 bp) sono stati visualizzati mediante elettroforesi su gel di agarosio e colorazione dell’acido nucleico verde brillante (eEnzima, Gaithersburg, MD, USA) per confermare la specificità del prodotto PCR.
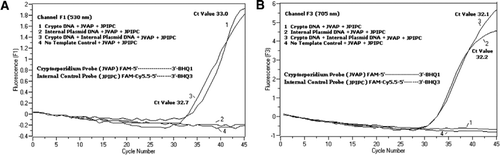
In tutte le miscele di reazione PCR è stata aggiunta una miscela equimolare di FAM/BHQ1 e FAM/Cy5.5/BHQ3. (A) I monitor del canale F1 aumentano la fluorescenza FAM per la presenza di C. parvum. Trama 1 (canale F1 raccoglie principalmente segnale FAM) contenente solo C. parvum DNA rivelato segnale FAM Cryptosporidium-specifico nella PCR. La trama 2 conteneva solo DNA plasmidico e nessun segnale FAM dalla sonda FAM/Cy5.5/BHQ3. Trama 3 conteneva sia C. parvum e plasmid DNA ma prodotto solo segnale FAM C. parvum-specifico. Il diagramma 4 era un campione negativo contenente solo acqua priva di nucleasi al posto di qualsiasi DNA modello; nessuna fluorescenza è stata osservata dalle sonde. (B) I monitor del canale F3 aumentano in Cy5.5 per la presenza di DNA plasmidico di controllo interno. Il diagramma 1 conteneva solo DNA di C. parvum e nessun segnale di fluorescenza dalla sonda FAM / BHQ1. Plot 2 (canale F3 raccoglie principalmente segnale Cy5.5) contenente solo DNA plasmidico rivelato controllo plasmidico interno (IPC)–specifica Cy5.5 segnale nella PCR. Trama 3 conteneva sia C. parvum e plasmid DNA ma prodotto solo IPC-specifica Cy5.5 segnale. Il diagramma 4 era un campione negativo contenente solo acqua libera da nucleasi al posto di qualsiasi DNA modello; nessuna fluorescenza è stata osservata dalle sonde.
Risultati e discussione
I segnali di fluorescenza PCR in tempo reale che riflettono l’amplificazione del DNA genomico di C. parvum sono stati rilevati nel canale F1 (Figura 2A) a causa dell’etichettatura FAM della sonda TaqMan. L’amplificazione del segnale a fluorescenza dell’IPC è stata rilevata nel canale F3 (Figura 2B) a causa di FAM/Cy5.5 etichettatura della sonda TaqMan IPC. Questa sonda ha prodotto un aumento di Cy5.5 nel canale F3 (Cy5.5), ma nessun aumento di FAM nel canale F1 (FAM) quando BHQ3 è separato dal FAM/Cy5.5 a causa dell’attività della nucleasi, perché l’energia è stata trasferita da FAM a Cy5.5. Allo stesso modo, nella reazione duplex, l’amplificazione del DNA genomico di C. parvum è stata rilevata nel canale F1, mentre l’amplificazione IPC è stata rilevata simultaneamente nel canale F3. FAM ha un’eccitazione a 495 nm e un’emissione a 520 nm. Cy5. 5 ha un’eccitazione di 690 nm e un’emissione di 705 nm. Con tale vicinanza tra Cy5.5 e FAM sulla sonda IPC a tripla etichetta, il fluoroforo Cy5.5 emette fluorescenza a causa del trasferimento completo di energia dal fluoroforo FAM, con conseguente assenza di emissione di fluorescenza di FAM nel canale F1 (Figura 2A). Le reazioni di controllo negative non hanno mostrato un aumento della fluorescenza a causa di FAM o Cy5.5. Questi risultati dimostrano anche che una singola sorgente LED blu (470 nm) è sufficiente per monitorare le emissioni a doppia fluorescenza attraverso i canali F1 (530 nm) e F3 (705 nm) senza sovrapposizioni spettrali nei rispettivi canali di monitoraggio per FAM e Cy5.5. La tecnica FRET-TaqMan è stata anche testata utilizzando un pannello di 12 estratti di DNA da campioni di feci (8 da campioni precedentemente determinati come positivi per Cryptosporidium spp. e quattro esemplari negativi al Cryptosporidium spp.). Il test 18S rRNA è stato in grado di rilevare tutti gli otto Cryptosporidium spp. Il test IPC TaqMan è stato trovato per amplificare solo il DNA plasmidico IPC. Il test IPC ha portato a valori Ct di 32-37, che indicavano fino a 5 valori Ct di inibizione della PCR per alcuni campioni di feci (32 era il valore Ct atteso per le reazioni senza inibizione).
La presente tecnica differisce dal TASTO tradizionalmente utilizzato in quanto viene utilizzata una sonda oligonucleotidica con etichetta fluorescente (sonda FRET-TaqMan) invece di due sonde (sonde FRET) in cui una sonda contiene il fluoroforo donatore e l’altra sonda contiene il fluoroforo accettore (13,14). La sonda FRET-TaqMan è etichettata all’estremità 5′ con due fluorofori di fluorofori donatore (FAM) e accettore (Cy5.5) e all’estremità 3′ con un quencher BHQ-3 come una sonda TaqMan. L’unica regione di legame della sonda è selezionata per differenziarsi dal DNA bersaglio in modo da monitorare due diverse misure di fluorescenza in due diversi canali di rilevamento (530 nm in F1 e 705 nm in F3) dalla stessa reazione di amplificazione senza la necessità di includere software di compensazione del colore. Il DNA bersaglio e l’IPC avevano gli stessi siti di legame del primer ma una sequenza di sonda diversa. Ciò assicura che l’uso degli stessi primer per entrambi gli obiettivi del DNA minimizzi la formazione del primer-dimero o l’interazione tra i set di primer. Quando i fluorofori FAM/Cy5.5 sono posti vicino all’estremità 5′, il trasferimento di energia di risonanza ha luogo con un aumento dell’emissione di fluorescenza del fluoroforo Cy5.5. Tuttavia, l’uso di BHQ3, un accettore di non fluorescenza per Cy5.5, diminuisce la fluorescenza di fondo potenziale, che si traduce in un rapporto segnale-rumore più elevato. Si è constatato che l’aggiunta di 100 copie del IPC ad una reazione contenente copies 100 copie di genomica C. parvum DNA ha portato a valori Ct simili (Ct = 32) quando il test Cryptosporidium TaqMan è stato eseguito come test singleplex e come test duplex con il test IPC TaqMan. Quando applicato a un pannello di campioni di DNA positivi al criptosporidio, l’IPC ha indicato che alcuni campioni non contenevano inibitori della PCR (basati su valori Ct attesi di 32), ma altri campioni erano associati a inibizione fino a 5 valori Ct.
Il controllo positivo PCR interno di nuova concezione si è dimostrato sensibile e specifico per il rilevamento simultaneo di due bersagli nella stessa reazione grazie all’etichettatura con il FAM/Cy5.5-BHQ3 e FAM/BHQ1 per il rispettivo gene bersaglio IPC e 18S rRNA in una PCR multiplex a due colori per il monitoraggio nei canali di fluorescenza F1 e F3 dello strumento R. A. P. I. D. L’uso della sonda etichettata 5’FAM-Cy5.5/3’BHQ-3 come secondo reporter è vantaggioso perché i massimi di emissione delle due sonde erano ben distanziati l’uno dall’altro. Il design della sonda a tripla etichetta FRET-TaqMan potrebbe avere applicazioni nello sviluppo di un dispositivo “lab-on-a-chip” monouso economico con ottica costituita da un’eccitazione a LED blu e due sorgenti di emissione di 530 nm e 705 nm, con filtri ottici appropriati per rilevare FAM in un canale e Cy5.5 nell’altro. Questa combinazione non richiederebbe due sorgenti di fluorescenza ad alta potenza e sistemi di rilevamento.
Riconoscimenti
Gli autori riconoscono l’assistenza di Mike Powers presso la struttura di sequenziamento del DNA e nucleo genomico (Università dello Utah, Salt Lake City, UT, USA) e Rachelle Muller presso Idaho Technology, Inc. per la discussione sullo sviluppo del controllo positivo interno per il ciclatore R. A. P. I. D. Questa pubblicazione è stata sostenuta in parte da fondi messi a disposizione attraverso i Centers for Disease Control and Prevention, Coordinating Office for Terrorism Preparedness and Emergency Response. L’uso di nomi commerciali e fonti commerciali è solo per l’identificazione e non implica l’approvazione da parte dei Centers for Disease Control and Prevention o del Dipartimento della Salute e dei servizi umani degli Stati Uniti.
- 1. Il suo nome deriva dal latino””. 2005. Sviluppo di un nuovo controllo positivo interno per i saggi basati su Taqman. Mol. Cellula. Sonde 19: 51-59.Crossref, Medline, CAS, Google Scholar
- 2. Il sito utilizza cookie tecnici e di terze parti per migliorare la tua esperienza di navigazione. 2004. Considerazioni pratiche nella progettazione di controlli di amplificazione interna per test diagnostici PCR. J. Clin. Microbiolo. 42:1863–1868.Crossref, Medline, CAS, Google Scholar
- 3. Il sito utilizza cookie tecnici e di terze parti. 2003. Costruzione e valutazione del DNA di controllo interno per PCR amplificazione del DNA di Chlamydia trachomatis da campioni di urina. J. Clin. Microbiolo. 41:1274–1276.Crossref, Medline, CAS, Google Scholar
- 4. Burggraf, S. e B. Olgemoller. 2004. Tecnica semplice per il controllo interno di test di amplificazione in tempo reale. Clin. Chimica. 50:819–825.Crossref, Medline, CAS, Google Scholar
- 5. Drosten, C., M. Weber, E. Seifried, e WK Roth. 2000. Valutazione di un nuovo test PCR con sequenza di controllo interno competitivo per lo screening dei donatori di sangue. Trasfusione 40: 718-724.Crossref, Medline, CAS, Google Scholar
- 6. Gruber, F., F. G. Falkner, F. Dorner, e T. Hammerle. 2001. Quantificazione del DNA virale mediante PCR in tempo reale mediante amplificazione duplex, standardizzazione interna e rilevazione di fluorescenza a due colori. Appl. Ambiente. Microbiolo. 67:2837–2839.Crossref, Medline, CAS, Google Scholar
- 7. Rosenstraus, M., Z. Il film è stato girato in Giappone e negli Stati Uniti. 1998. Un controllo interno per la PCR diagnostica di routine: progettazione, proprietà ed effetto sulle prestazioni cliniche. J. Clin. Microbiolo. 36:191–197.Medline, CAS, Google Scholar
- 8. Stocher, M., V. Leb, e J. Berg. 2003. Un approccio conveniente alla generazione di DNA di controllo interno multiplo per un pannello di analisi PCR in tempo reale. J. Virol. Metodi 108:1-8.Crossref, Medline, CAS, Google Scholar
- 9. Zimmermann, K. e J. W. Mannhalter. 1996. Aspetti tecnici della PCR competitiva quantitativa. BioTechniques 21:268-279.Link, CAS, Google Scholar
- 10. Jothikumar, N., A. J. da Silva, I. Moura, Y. Qvarnstrom, e V. R. Hill. 2008. Rilevamento e differenziazione di Cryptosporidium hominis e Cryptosporidium parvum mediante doppio test TaqMan. J. Med. Microbiolo. 57:1099–1105.Crossref, Medline, CAS, Google Scholar
- 11. Il sito utilizza cookie tecnici e di terze parti. 1990. Strumento di ricerca di allineamento locale di base. J. Mol. Biol. 215:403–410.Crossref, Medline, CAS, Google Scholar
- 12. Il suo nome deriva dal greco antico, che significa “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”. 2007. Valutazione multistatale di una procedura basata sull’ultrafiltrazione per il recupero simultaneo di microbi enterici in campioni di acqua di rubinetto da 100 litri. Appl. Ambiente. Microbiolo. 73:4218–4225.Crossref, Medline, CAS, Google Scholar
- 13. Bernard, P.S., M. J. Lay, e CT Wittwer. 1998. Amplificazione integrata e rilevazione della mutazione del punto C677T nel gene della metilenetetraidrofolato reduttasi mediante trasferimento di energia di risonanza a fluorescenza e curve di fusione della sonda. Anale. Biochimica. 255:101–107.Crossref, Medline, CAS, Google Scholar
- 14. Wittwer, C. T., M. G. Herrmann, A. A. Moss, e R. P. Rasmussen. 1997. Continuous fluorescence monitoring of rapid cycle DNA amplification. BioTechniques 22:130–138.Link, CAS, Google Scholar